SDGs
連携提案
この研究室では、化学の標準であるガウス型基底関数による密度汎関数法に基づき、タンパク質の正準分子軌道計算プログラムProteinDF/QCLObotを研究開発している。ソフトウエアはhttps://proteindf.github.ioにて公開中である。
この方法を用いると、例えば、図1のように古典計算とは明らかに差異のある精密な静電ポテンシャルや、図2のように機能を反映した分子軌道がタンパク質で計算できる。これらを解析することで、どのアミノ酸残基がそのタンパク質の物性に影響を与えているのかを電子レベルで解析することができる。
基礎研究のみならず、新規ペプチド・酵素設計といった応用研究で威力を発揮し、有意義な共同研究が実施できるものと考えられる。
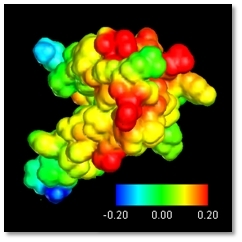
図1 インスリン静電ポテンシャルの全電子計算と古典計算の差
主流の古典計算結果では、misleadingを招く恐れがある。
© 佐藤文俊
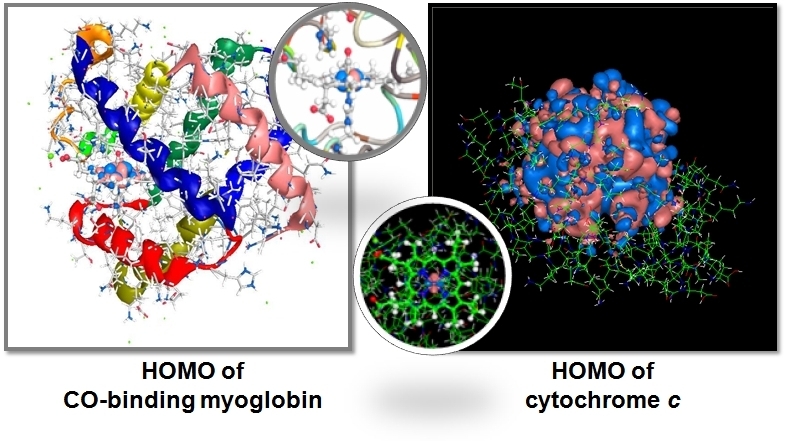
図2 ミオグロビンとシトクロムcのHOMOの比較
同じヘムタンパク質でも機能により全く電子状態が異なる。全電子計算ならではの結果。
© 佐藤文俊

















